Numpy: producto cartesiano de puntos de matriz x e y en matriz única de puntos 2D
Tengo dos matrices numpy que definen los ejes x e y de una cuadrícula. Por ejemplo:
x = numpy.array([1,2,3])
y = numpy.array([4,5])
Me gustaría generar el producto cartesiano de estos arrays para generar:
array([[1,4],[2,4],[3,4],[1,5],[2,5],[3,5]])
De una manera que no es terriblemente ineficiente ya que necesito hacer esto muchas veces en un bucle. Asumo que convertirlos a una lista de Python y usar itertools.product y volver a una matriz numpy no es la forma más eficiente.
11 answers
>>> numpy.transpose([numpy.tile(x, len(y)), numpy.repeat(y, len(x))])
array([[1, 4],
[2, 4],
[3, 4],
[1, 5],
[2, 5],
[3, 5]])
Ver Usando numpy para construir una matriz de todas las combinaciones de dos matrices para una solución general para calcular el producto cartesiano de N matrices.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2017-05-23 11:55:10
Un canónico cartesian_product (casi)
Hay muchos enfoques para este problema con diferentes propiedades. Algunos son más rápidos que otros, y algunos son de propósito más general. Después de muchas pruebas y ajustes, he encontrado que la siguiente función, que calcula un cartesian_product n-dimensional, es más rápida que la mayoría de las otras para muchas entradas. Para un par de enfoques que son un poco más complejos, pero son incluso un poco más rápidos en muchos casos, ver la respuesta de Paul Panzer .
Dada esa respuesta, esta ya no es la implementación más rápida del producto cartesiano en numpy que yo sepa. Sin embargo, creo que su simplicidad seguirá siendo un punto de referencia útil para futuras mejoras:
def cartesian_product(*arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
Vale la pena mencionar que esta función usa ix_ de una manera inusual; mientras que el uso documentado de ix_ es para generar índices en una matriz, sucede que las matrices con la misma forma se puede utilizar para la asignación transmitida. Muchas gracias a mgilson , quien me inspiró a intentar usar ix_de esta manera, y a unutbu , quien proporcionó algunos comentarios extremadamente útiles sobre esta respuesta, incluida la sugerencia de usar numpy.result_type.
Alternativas notables
A veces es más rápido escribir bloques de memoria contiguos en orden Fortran. Esa es la base de esta alternativa, cartesian_product_transpose, que ha demostrado ser más rápida en algunos hardware que cartesian_product (ver más abajo). Sin embargo, la respuesta de Paul Panzer, que utiliza el mismo principio, es aún más rápida. Aún así, incluyo esto aquí para los lectores interesados:
def cartesian_product_transpose(*arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
Después de entender el enfoque de Panzer, escribí una nueva versión que es casi tan rápida como la suya, y es casi tan simple como cartesian_product:
def cartesian_product_simple_transpose(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([la] + [len(a) for a in arrays], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[i, ...] = a
return arr.reshape(la, -1).T
Esto parece tener una sobrecarga de tiempo constante que hace que funcione más lento que el Panzer para entradas pequeñas. Pero para entradas más grandes, en todas las pruebas que realicé, funciona tan bien como su más rápido aplicación (cartesian_product_transpose_pp).
En las siguientes secciones, incluyo algunas pruebas de otras alternativas. Estos ahora están un poco anticuados, pero en lugar de duplicar el esfuerzo, he decidido dejarlos aquí por interés histórico. Para pruebas actualizadas, ver la respuesta de Panzer, así como Nico Schlömer 's.
Pruebas con alternativas
Aquí hay una batería de pruebas que muestran el aumento de rendimiento que algunas de estas funciones proporcionan en relación con un número de alternativa. Todas las pruebas que se muestran aquí se realizaron en una máquina de cuatro núcleos, ejecutando Mac OS 10.12.5, Python 3.6.1 y numpy 1.12.1. Se sabe que las variaciones en el hardware y el software producen resultados diferentes, por lo que YMMV. Ejecutar estas pruebas por ti mismo para estar seguro!
Definiciones:
import numpy
import itertools
from functools import reduce
### Two-dimensional products ###
def repeat_product(x, y):
return numpy.transpose([numpy.tile(x, len(y)),
numpy.repeat(y, len(x))])
def dstack_product(x, y):
return numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
### Generalized N-dimensional products ###
def cartesian_product(*arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(*arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
# from https://stackoverflow.com/a/1235363/577088
def cartesian_product_recursive(*arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:,0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m,1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m,1:] = out[0:m,1:]
return out
def cartesian_product_itertools(*arrays):
return numpy.array(list(itertools.product(*arrays)))
### Test code ###
name_func = [('repeat_product',
repeat_product),
('dstack_product',
dstack_product),
('cartesian_product',
cartesian_product),
('cartesian_product_transpose',
cartesian_product_transpose),
('cartesian_product_recursive',
cartesian_product_recursive),
('cartesian_product_itertools',
cartesian_product_itertools)]
def test(in_arrays, test_funcs):
global func
global arrays
arrays = in_arrays
for name, func in test_funcs:
print('{}:'.format(name))
%timeit func(*arrays)
def test_all(*in_arrays):
test(in_arrays, name_func)
# `cartesian_product_recursive` throws an
# unexpected error when used on more than
# two input arrays, so for now I've removed
# it from these tests.
def test_cartesian(*in_arrays):
test(in_arrays, name_func[2:4] + name_func[-1:])
x10 = [numpy.arange(10)]
x50 = [numpy.arange(50)]
x100 = [numpy.arange(100)]
x500 = [numpy.arange(500)]
x1000 = [numpy.arange(1000)]
Resultados de la prueba:
In [2]: test_all(*(x100 * 2))
repeat_product:
67.5 µs ± 633 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
dstack_product:
67.7 µs ± 1.09 µs per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product:
33.4 µs ± 558 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product_transpose:
67.7 µs ± 932 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product_recursive:
215 µs ± 6.01 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_itertools:
3.65 ms ± 38.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
In [3]: test_all(*(x500 * 2))
repeat_product:
1.31 ms ± 9.28 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
dstack_product:
1.27 ms ± 7.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product:
375 µs ± 4.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_transpose:
488 µs ± 8.88 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_recursive:
2.21 ms ± 38.4 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
105 ms ± 1.17 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [4]: test_all(*(x1000 * 2))
repeat_product:
10.2 ms ± 132 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
dstack_product:
12 ms ± 120 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product:
4.75 ms ± 57.1 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_transpose:
7.76 ms ± 52.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_recursive:
13 ms ± 209 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
422 ms ± 7.77 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
En todos los casos, cartesian_product como se define al principio de esta respuesta es más rápido.
Para aquellas funciones que aceptan un número arbitrario de entradas arrays, vale la pena comprobar el rendimiento cuando len(arrays) > 2 también. (Hasta que pueda determinar por qué cartesian_product_recursive lanza un error en este caso, lo he eliminado de estas pruebas.)
In [5]: test_cartesian(*(x100 * 3))
cartesian_product:
8.8 ms ± 138 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_transpose:
7.87 ms ± 91.5 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
518 ms ± 5.5 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [6]: test_cartesian(*(x50 * 4))
cartesian_product:
169 ms ± 5.1 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_transpose:
184 ms ± 4.32 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_itertools:
3.69 s ± 73.5 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [7]: test_cartesian(*(x10 * 6))
cartesian_product:
26.5 ms ± 449 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_transpose:
16 ms ± 133 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
728 ms ± 16 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [8]: test_cartesian(*(x10 * 7))
cartesian_product:
650 ms ± 8.14 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
cartesian_product_transpose:
518 ms ± 7.09 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
cartesian_product_itertools:
8.13 s ± 122 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Como muestran estas pruebas, cartesian_product sigue siendo competitivo hasta que el número de matrices de entrada se eleva por encima (aproximadamente) de cuatro. Después de eso, cartesian_product_transpose tiene un ligero borde.
Vale la pena reiterar que los usuarios con otro hardware y sistemas operativos pueden ver resultados diferentes. Por ejemplo, unutbu informa que los siguientes resultados para estas pruebas usando Ubuntu 14.04, Python 3.4.3, y numpy 1.14.0.dev0 + b7050a9:
>>> %timeit cartesian_product_transpose(x500, y500)
1000 loops, best of 3: 682 µs per loop
>>> %timeit cartesian_product(x500, y500)
1000 loops, best of 3: 1.55 ms per loop
A continuación, entro en algunos detalles sobre las pruebas anteriores que he realizado a lo largo de estas líneas. El rendimiento relativo de estos enfoques ha cambiado con el tiempo, para diferentes hardware y diferentes versiones de Python y numpy. Si bien no es inmediatamente útil para las personas que usan versiones actualizadas de numpy, ilustra cómo han cambiado las cosas desde la primera versión de este respuesta.
Una alternativa simple: meshgrid + dstack
La respuesta actualmente aceptada usa tile y repeat para transmitir dos matrices juntas. Pero la función meshgrid hace prácticamente lo mismo. Aquí está la salida de tile y repeat antes de pasar a transponer:
In [1]: import numpy
In [2]: x = numpy.array([1,2,3])
...: y = numpy.array([4,5])
...:
In [3]: [numpy.tile(x, len(y)), numpy.repeat(y, len(x))]
Out[3]: [array([1, 2, 3, 1, 2, 3]), array([4, 4, 4, 5, 5, 5])]
Y aquí está la salida de meshgrid:
In [4]: numpy.meshgrid(x, y)
Out[4]:
[array([[1, 2, 3],
[1, 2, 3]]), array([[4, 4, 4],
[5, 5, 5]])]
Como pueden ver, es casi idéntico. Solo necesitamos remodelar el resultado para obtener exactamente el mismo resultado.
In [5]: xt, xr = numpy.meshgrid(x, y)
...: [xt.ravel(), xr.ravel()]
Out[5]: [array([1, 2, 3, 1, 2, 3]), array([4, 4, 4, 5, 5, 5])]
En lugar de remodelando en este punto, sin embargo, podríamos pasar la salida de meshgrid a dstack y remodelar después, lo que ahorra algo de trabajo:
In [6]: numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
Out[6]:
array([[1, 4],
[2, 4],
[3, 4],
[1, 5],
[2, 5],
[3, 5]])
Contrariamente a la afirmación en este comentario, no he visto evidencia de que diferentes entradas produzcan salidas de forma diferente, y como lo demuestra lo anterior, hacen cosas muy similares, por lo que sería bastante extraño si lo hicieran. Por favor, hágamelo saber si encuentra un contraejemplo.
Pruebas meshgrid + dstack vs repeat + transpose
El desempeño relativo de estos dos enfoques ha cambiado con el tiempo. En una versión anterior de Python (2.7), el resultado usando meshgrid + dstack era notablemente más rápido para pequeñas entradas. (Tenga en cuenta que estas pruebas son de una versión antigua de esta respuesta. Definiciones:
>>> def repeat_product(x, y):
... return numpy.transpose([numpy.tile(x, len(y)),
numpy.repeat(y, len(x))])
...
>>> def dstack_product(x, y):
... return numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
...
Para la entrada de tamaño moderado, vi una aceleración significativa. Pero volví a probar estas pruebas con versiones más recientes de Python (3.6.1) y numpy (1.12.1), en una máquina más nueva. Los dos enfoques son casi idéntico ahora.
Prueba antigua
>>> x, y = numpy.arange(500), numpy.arange(500)
>>> %timeit repeat_product(x, y)
10 loops, best of 3: 62 ms per loop
>>> %timeit dstack_product(x, y)
100 loops, best of 3: 12.2 ms per loop
Nueva Prueba
In [7]: x, y = numpy.arange(500), numpy.arange(500)
In [8]: %timeit repeat_product(x, y)
1.32 ms ± 24.7 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
In [9]: %timeit dstack_product(x, y)
1.26 ms ± 8.47 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
Como siempre, YMMV, pero esto sugiere que en versiones recientes de Python y numpy, estos son intercambiables.
Funciones generalizadas del producto
En general, podríamos esperar que el uso de funciones integradas sea más rápido para entradas pequeñas, mientras que para entradas grandes, una función especialmente diseñada podría ser más rápida. Además para un producto n-dimensional generalizado, tile y repeat no ayudarán, porque no tienen análogos claros de dimensiones superiores. Por lo tanto, vale la pena investigar el comportamiento de las funciones diseñadas específicamente.
La mayoría de las pruebas relevantes aparecen al principio de esta respuesta, pero aquí están algunas de las pruebas realizadas en versiones anteriores de Python y numpy para la comparación.
La función cartesian definida en otra respuesta solía funcionar bastante bien para entradas más grandes. (Es lo mismo que la función llamado cartesian_product_recursive arriba.) Para comparar cartesian con dstack_prodct, usamos solo dos dimensiones.
Aquí de nuevo, la antigua prueba mostró una diferencia significativa, mientras que la nueva prueba muestra casi ninguna.
Prueba antigua
>>> x, y = numpy.arange(1000), numpy.arange(1000)
>>> %timeit cartesian([x, y])
10 loops, best of 3: 25.4 ms per loop
>>> %timeit dstack_product(x, y)
10 loops, best of 3: 66.6 ms per loop
Nueva Prueba
In [10]: x, y = numpy.arange(1000), numpy.arange(1000)
In [11]: %timeit cartesian([x, y])
12.1 ms ± 199 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
In [12]: %timeit dstack_product(x, y)
12.7 ms ± 334 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
Como antes, dstack_product todavía late cartesian a escalas más pequeñas.
Nueva Prueba (prueba anterior redundante no mostrada )
In [13]: x, y = numpy.arange(100), numpy.arange(100)
In [14]: %timeit cartesian([x, y])
215 µs ± 4.75 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
In [15]: %timeit dstack_product(x, y)
65.7 µs ± 1.15 µs per loop (mean ± std. dev. of 7 runs, 10000 loops each)
Estas distinciones son, creo, interesantes y vale la pena grabarlo; pero al final son académicos. Como mostraron las pruebas al principio de esta respuesta, todas estas versiones son casi siempre más lentas que cartesian_product, definidas al principio de esta respuesta which que es en sí un poco más lenta que las implementaciones más rápidas entre las respuestas a esta pregunta.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-03-28 15:31:59
Solo puedes hacer una comprensión de lista normal en python
x = numpy.array([1,2,3])
y = numpy.array([4,5])
[[x0, y0] for x0 in x for y0 in y]
Que debería darte
[[1, 4], [1, 5], [2, 4], [2, 5], [3, 4], [3, 5]]
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2013-10-18 22:00:34
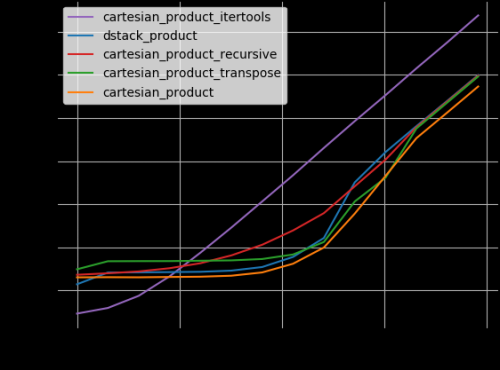
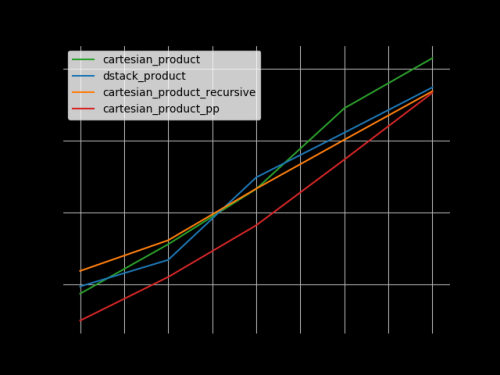
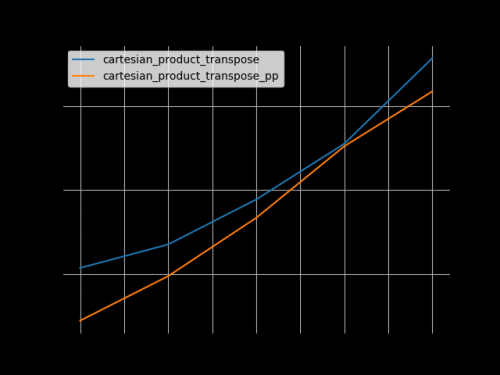
También estaba interesado en esto e hice una pequeña comparación de rendimiento, quizás algo más clara que en la respuesta de @senderle.
Para dos matrices (el caso clásico):
Para cuatro matrices:
(Tenga en cuenta que la longitud de los arrays es solo unas pocas docenas de entradas aquí.)
Código para reproducir las parcelas:
from functools import reduce
import itertools
import numpy
import perfplot
def dstack_product(arrays):
return numpy.dstack(
numpy.meshgrid(*arrays, indexing='ij')
).reshape(-1, len(arrays))
# Generalized N-dimensional products
def cartesian_product(arrays):
la = len(arrays)
dtype = numpy.find_common_type([a.dtype for a in arrays], [])
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[..., i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = reduce(numpy.multiply, broadcasted[0].shape), len(broadcasted)
dtype = numpy.find_common_type([a.dtype for a in arrays], [])
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
# from https://stackoverflow.com/a/1235363/577088
def cartesian_product_recursive(arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:, 0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m, 1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m, 1:] = out[0:m, 1:]
return out
def cartesian_product_itertools(arrays):
return numpy.array(list(itertools.product(*arrays)))
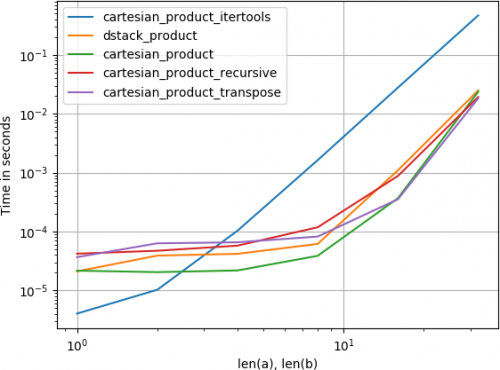
perfplot.show(
setup=lambda n: 4*(numpy.arange(n, dtype=float),),
n_range=[2**k for k in range(6)],
kernels=[
dstack_product,
cartesian_product,
cartesian_product_transpose,
cartesian_product_recursive,
cartesian_product_itertools
],
logx=True,
logy=True,
xlabel='len(a), len(b)',
equality_check=None
)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-06-12 10:23:59
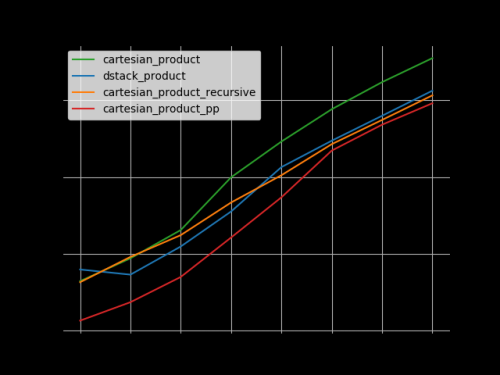
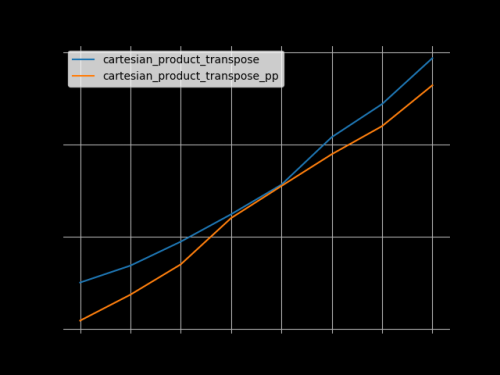
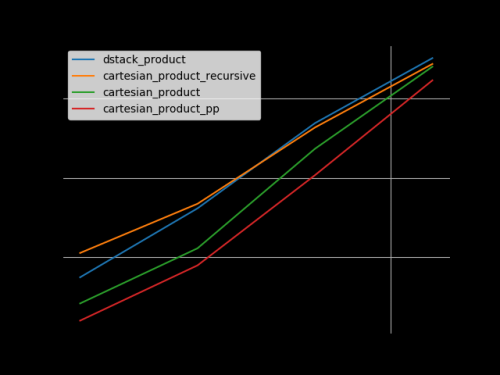
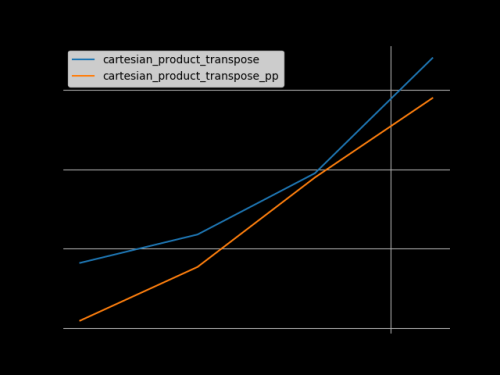
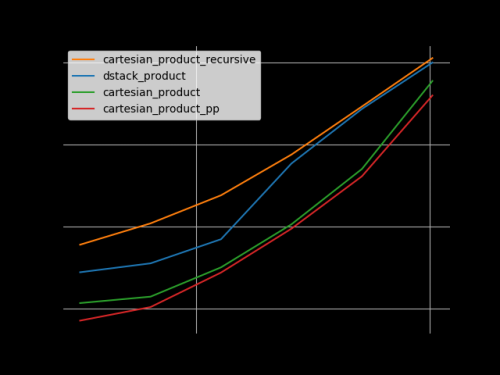
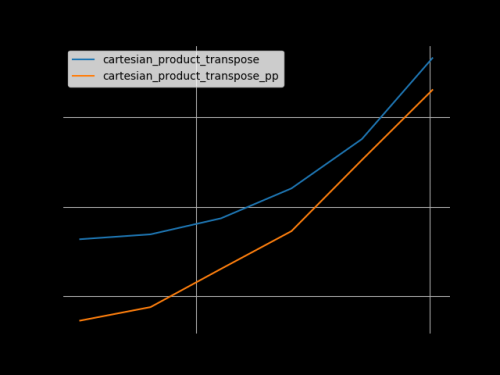
Basándose en el trabajo de base ejemplar de @senderle, se me han ocurrido dos versiones, una para C y otra para Fortran, que a menudo son un poco más rápidas.
-
cartesian_product_transpose_ppes - a diferencia decartesian_product_transposede @senderle que usa una estrategia completamente diferente - una versión decartesion_productque usa el diseño de memoria de transposición más favorable + algunas optimizaciones muy menores. -
cartesian_product_ppse adhiere al diseño de memoria original. Lo que lo hace rápido es su uso de copia contigua. Las copias contiguas resultan en ser mucho más rápido que copiar un bloque completo de memoria a pesar de que solo una parte de ella contiene datos válidos es preferible a copiar solo los bits válidos.
Algunos perfplots. Hice diseños separados para C y Fortran, porque estas son tareas diferentes IMO.
Los nombres que terminan en 'pp' son mis enfoques.
1) muchos factores diminutos (2 elementos cada uno)
2) muchos factores pequeños (4 elementos cada uno)
3) tres factores de igual longitud
4) dos factores de igual longitud
Código (necesidad de hacer corridas separadas para cada parcela b / c No pude averiguar cómo restablecer; también necesidad de editar / comentar in / out apropiadamente):
import numpy
import numpy as np
from functools import reduce
import itertools
import timeit
import perfplot
def dstack_product(arrays):
return numpy.dstack(
numpy.meshgrid(*arrays, indexing='ij')
).reshape(-1, len(arrays))
def cartesian_product_transpose_pp(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty((la, *map(len, arrays)), dtype=dtype)
idx = slice(None), *itertools.repeat(None, la)
for i, a in enumerate(arrays):
arr[i, ...] = a[idx[:la-i]]
return arr.reshape(la, -1).T
def cartesian_product(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
from itertools import accumulate, repeat, chain
def cartesian_product_pp(arrays, out=None):
la = len(arrays)
L = *map(len, arrays), la
dtype = numpy.result_type(*arrays)
arr = numpy.empty(L, dtype=dtype)
arrs = *accumulate(chain((arr,), repeat(0, la-1)), np.ndarray.__getitem__),
idx = slice(None), *itertools.repeat(None, la-1)
for i in range(la-1, 0, -1):
arrs[i][..., i] = arrays[i][idx[:la-i]]
arrs[i-1][1:] = arrs[i]
arr[..., 0] = arrays[0][idx]
return arr.reshape(-1, la)
def cartesian_product_itertools(arrays):
return numpy.array(list(itertools.product(*arrays)))
# from https://stackoverflow.com/a/1235363/577088
def cartesian_product_recursive(arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:, 0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m, 1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m, 1:] = out[0:m, 1:]
return out
### Test code ###
if False:
perfplot.save('cp_4el_high.png',
setup=lambda n: n*(numpy.arange(4, dtype=float),),
n_range=list(range(6, 11)),
kernels=[
dstack_product,
cartesian_product_recursive,
cartesian_product,
# cartesian_product_transpose,
cartesian_product_pp,
# cartesian_product_transpose_pp,
],
logx=False,
logy=True,
xlabel='#factors',
equality_check=None
)
else:
perfplot.save('cp_2f_T.png',
setup=lambda n: 2*(numpy.arange(n, dtype=float),),
n_range=[2**k for k in range(5, 11)],
kernels=[
# dstack_product,
# cartesian_product_recursive,
# cartesian_product,
cartesian_product_transpose,
# cartesian_product_pp,
cartesian_product_transpose_pp,
],
logx=True,
logy=True,
xlabel='length of each factor',
equality_check=None
)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-03-29 17:40:56
A partir de octubre. 2017, numpy ahora tiene una función genérica np.stack que toma un parámetro axis. Usándolo, podemos tener un "producto cartesiano generalizado " usando la técnica" dstack y meshgrid":
import numpy as np
def cartesian_product(*arrays):
ndim = len(arrays)
return np.stack(np.meshgrid(*arrays), axis=-1).reshape(-1, ndim)
Nota sobre el parámetro axis=-1. Este es el último eje (más interno) en el resultado. Es equivalente a usar axis=ndim.
Otro comentario, ya que los productos cartesianos explotan muy rápidamente, a menos que necesitemos realizar la matriz en memoria por alguna razón, si el producto es muy grande, es posible que desee hacer uso de itertools y utilizar los valores sobre la marcha.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-03-30 01:54:24
Usé @kennytm answer por un tiempo, pero al intentar hacer lo mismo en TensorFlow, pero encontré que TensorFlow no tiene equivalente a numpy.repeat(). Después de un poco de experimentación, creo que encontré una solución más general para vectores arbitrarios de puntos.
Para numpy:
import numpy as np
def cartesian_product(*args: np.ndarray) -> np.ndarray:
"""
Produce the cartesian product of arbitrary length vectors.
Parameters
----------
np.ndarray args
vector of points of interest in each dimension
Returns
-------
np.ndarray
the cartesian product of size [m x n] wherein:
m = prod([len(a) for a in args])
n = len(args)
"""
for i, a in enumerate(args):
assert a.ndim == 1, "arg {:d} is not rank 1".format(i)
return np.concatenate([np.reshape(xi, [-1, 1]) for xi in np.meshgrid(*args)], axis=1)
Y para TensorFlow:
import tensorflow as tf
def cartesian_product(*args: tf.Tensor) -> tf.Tensor:
"""
Produce the cartesian product of arbitrary length vectors.
Parameters
----------
tf.Tensor args
vector of points of interest in each dimension
Returns
-------
tf.Tensor
the cartesian product of size [m x n] wherein:
m = prod([len(a) for a in args])
n = len(args)
"""
for i, a in enumerate(args):
tf.assert_rank(a, 1, message="arg {:d} is not rank 1".format(i))
return tf.concat([tf.reshape(xi, [-1, 1]) for xi in tf.meshgrid(*args)], axis=1)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-02-20 16:27:52
El paquete Scikit-learn tiene una implementación rápida de exactamente esto:
from sklearn.utils.extmath import cartesian
product = cartesian((x,y))
Tenga en cuenta que la convención de esta implementación es diferente de lo que desea, si se preocupa por el orden de la salida. Para su pedido exacto, puede hacer
product = cartesian((y,x))[:, ::-1]
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-03-26 19:48:19
Más generalmente, si tiene dos matrices numpy 2d a y b, y desea concatenar cada fila de a a cada fila de b (Un producto cartesiano de filas, como una combinación en una base de datos), puede usar este método:
import numpy
def join_2d(a, b):
assert a.dtype == b.dtype
a_part = numpy.tile(a, (len(b), 1))
b_part = numpy.repeat(b, len(a), axis=0)
return numpy.hstack((a_part, b_part))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2016-01-05 11:38:32
Lo más rápido que puede obtener es combinar una expresión generadora con la función map:
import numpy
import datetime
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = (item for sublist in [list(map(lambda x: (x,i),a)) for i in b] for item in sublist)
print (list(foo))
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Salidas (en realidad se imprime toda la lista resultante):
[(0, 0), (1, 0), ...,(998, 199), (999, 199)]
execution time: 1.253567 s
O usando una expresión de doble generador:
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = ((x,y) for x in a for y in b)
print (list(foo))
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Salidas (lista completa impresa):
[(0, 0), (1, 0), ...,(998, 199), (999, 199)]
execution time: 1.187415 s
Tenga en cuenta que la mayor parte del tiempo de cálculo va en el comando de impresión. Los cálculos del generador son decentemente eficientes. Sin imprimir los tiempos de cálculo son:
execution time: 0.079208 s
Para la expresión del generador + función de mapa y:
execution time: 0.007093 s
Para la expresión double generator.
Si lo que realmente desea es calcular el producto real de cada uno de los pares de coordenadas, lo más rápido es resolverlo como un producto de matriz numpy:
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = np.dot(np.asmatrix([[i,0] for i in a]), np.asmatrix([[i,0] for i in b]).T)
print (foo)
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Salidas:
[[ 0 0 0 ..., 0 0 0]
[ 0 1 2 ..., 197 198 199]
[ 0 2 4 ..., 394 396 398]
...,
[ 0 997 1994 ..., 196409 197406 198403]
[ 0 998 1996 ..., 196606 197604 198602]
[ 0 999 1998 ..., 196803 197802 198801]]
execution time: 0.003869 s
Y sin imprimir (en este caso no ahorra mucho ya que solo se imprime una pequeña parte de la matriz):
execution time: 0.003083 s
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-04-24 14:05:36
Esto también se puede hacer fácilmente usando itertools.método del producto
from itertools import product
import numpy as np
x = np.array([1, 2, 3])
y = np.array([4, 5])
cart_prod = np.array(list(product(*[[x], [y]])),dtype='int32')
Resultado:
matriz([
[1, 4],
[1, 5],
[2, 4],
[2, 5],
[3, 4],
[3, 5]], dtype = int32)
Tiempo de Ejecución: 0.000155 s
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2018-03-29 13:42:57