Enfoque sencillo para asignar clústeres para nuevos datos después de la agrupación en clústeres de k-means
Estoy ejecutando k-means clustering en un marco de datos df1, y estoy buscando un enfoque simple para calcular el centro de clúster más cercano para cada observación en un nuevo marco de datos df2 (con los mismos nombres de variable). Piense en df1 como el conjunto de entrenamiento y df2 en el conjunto de pruebas; quiero agrupar en el conjunto de entrenamiento y asignar cada punto de prueba al clúster correcto.
Sé cómo hacer esto con la función apply y algunas funciones simples definidas por el usuario (publicaciones anteriores sobre el tema tienen por lo general propuso algo similar):
df1 <- data.frame(x=runif(100), y=runif(100))
df2 <- data.frame(x=runif(100), y=runif(100))
km <- kmeans(df1, centers=3)
closest.cluster <- function(x) {
cluster.dist <- apply(km$centers, 1, function(y) sqrt(sum((x-y)^2)))
return(which.min(cluster.dist)[1])
}
clusters2 <- apply(df2, 1, closest.cluster)
Sin embargo, estoy preparando este ejemplo de clústeres para un curso en el que los estudiantes no estarán familiarizados con la función apply, por lo que preferiría mucho si pudiera asignar los clústeres a df2 con una función incorporada. ¿Hay alguna función incorporada conveniente para encontrar el clúster más cercano?
2 answers
Podría usar el paquete flexclust , que tiene un método predict implementado para k-means:
library("flexclust")
data("Nclus")
set.seed(1)
dat <- as.data.frame(Nclus)
ind <- sample(nrow(dat), 50)
dat[["train"]] <- TRUE
dat[["train"]][ind] <- FALSE
cl1 = kcca(dat[dat[["train"]]==TRUE, 1:2], k=4, kccaFamily("kmeans"))
cl1
#
# call:
# kcca(x = dat[dat[["train"]] == TRUE, 1:2], k = 4)
#
# cluster sizes:
#
# 1 2 3 4
#130 181 98 91
pred_train <- predict(cl1)
pred_test <- predict(cl1, newdata=dat[dat[["train"]]==FALSE, 1:2])
image(cl1)
points(dat[dat[["train"]]==TRUE, 1:2], col=pred_train, pch=19, cex=0.3)
points(dat[dat[["train"]]==FALSE, 1:2], col=pred_test, pch=22, bg="orange")
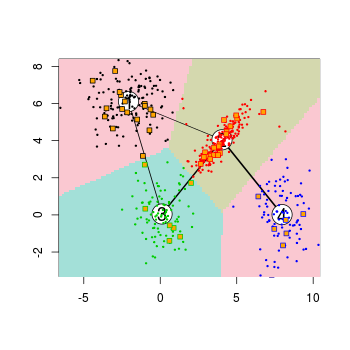
También hay métodos de conversión para convertir los resultados de funciones de clúster como stats::kmeans o cluster::pam a objetos de clase kcca y viceversa:
as.kcca(cl, data=x)
# kcca object of family ‘kmeans’
#
# call:
# as.kcca(object = cl, data = x)
#
# cluster sizes:
#
# 1 2
# 50 50
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2013-12-16 22:04:34
Algo que noté sobre el enfoque en la pregunta y los enfoques flexclust es que son bastante lentos (benchmarked aquí para un conjunto de entrenamiento y pruebas con 1 millón de observaciones con 2 características cada uno).
El ajuste del modelo original es razonablemente rápido:
set.seed(144)
df1 <- data.frame(x=runif(1e6), y=runif(1e6))
df2 <- data.frame(x=runif(1e6), y=runif(1e6))
system.time(km <- kmeans(df1, centers=3))
# user system elapsed
# 1.204 0.077 1.295
La solución que publiqué en la pregunta es lenta en el cálculo de las asignaciones de clúster de conjunto de prueba, ya que llama por separado closest.cluster para cada punto de conjunto de prueba:
system.time(pred.test <- apply(df2, 1, closest.cluster))
# user system elapsed
# 42.064 0.251 42.586
Mientras tanto, el flexclust el paquete parece agregar una gran cantidad de sobrecarga independientemente de si convertimos el modelo ajustado con as.kcca o ajustamos uno nuevo nosotros mismos con kcca (aunque la predicción al final es mucho más rápida)
# APPROACH #1: Convert from the kmeans() output
system.time(km.flexclust <- as.kcca(km, data=df1))
# user system elapsed
# 87.562 1.216 89.495
system.time(pred.flexclust <- predict(km.flexclust, newdata=df2))
# user system elapsed
# 0.182 0.065 0.250
# Approach #2: Fit the k-means clustering model in the flexclust package
system.time(km.flexclust2 <- kcca(df1, k=3, kccaFamily("kmeans")))
# user system elapsed
# 125.193 7.182 133.519
system.time(pred.flexclust2 <- predict(km.flexclust2, newdata=df2))
# user system elapsed
# 0.198 0.084 0.302
Parece que hay otro enfoque sensato aquí: usar una solución rápida de k-vecinos más cercanos como un árbol k-d para encontrar el vecino más cercano de cada observación de conjunto de pruebas dentro del conjunto de centroides del clúster. Esto se puede escribir de forma compacta y es relativamente rápido:
library(FNN)
system.time(pred.knn <- get.knnx(km$center, df2, 1)$nn.index[,1])
# user system elapsed
# 0.315 0.013 0.345
all(pred.test == pred.knn)
# [1] TRUE
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2017-04-25 14:43:15